DNA-এর ডাবল-হেলিক্স কাঠামোর উন্মোচনের পরপরই ওয়াটসন ও ক্রিক এই DNA-এর সম্ভাব্য প্রতিলিপি বা রেপ্লিকেশনের পদ্ধতি সম্পর্কে ধারণা দেন। তাদের এই ধারণাটি অর্ধ-রক্ষণশীল বা সেমি-কন্সার্ভেটিভ (Semi conservative) মডেল নামে সুপরিচিত। তাদের সমসাময়িক আরো দুটি ধারণা ছিলো, রক্ষণশীল-প্রতিলিপি বা কন্সার্ভেটিভ-রেপ্লিকেশন (Conservative replication) মডেল এবং এলোমেলো-প্রতিলিপি বা ডেসপারর্সিভ-রেপ্লিকেশন (Despersive replication) মডেল। কন্সার্ভেটিভ মডেলে আদি DNA অক্ষত থাকে এবং এথেকে সম্পুর্ন নতুন DNA তৈরি করা হয়। ডেসপারর্সিভ মডেলে আদি DNA-কে ছোট ছোট টুকরোয় কেঁটে ফেলা হয়। এই ছোট ছোট টুকরোগুলো তাদের কমপ্লিমেন্টারী বা পরিপূরক নিউক্লিয়টাইডের সাথে যুক্ত হবার মাধ্যমে দুটি নতুন DNA তৈরি করে। এই প্রক্রিয়ার আদি DNA-টি হারিয়ে যায়। অন্যদিকে, সেমি-কন্সার্ভেটিভ মডেল অনুসারে, রেপ্লিকেশন প্রক্রিয়ায় প্রথমে DNA-এর কমপ্লিমেন্টারি স্ট্র্যান্ড দুটি আলাদা হয়ে যায়, এরপর এই আলাদা আলাদা স্ট্র্যান্ডগুলো তাদের নতুন কমপ্লিমেন্টারি স্ট্র্যান্ড তৈরি করে নেয়।
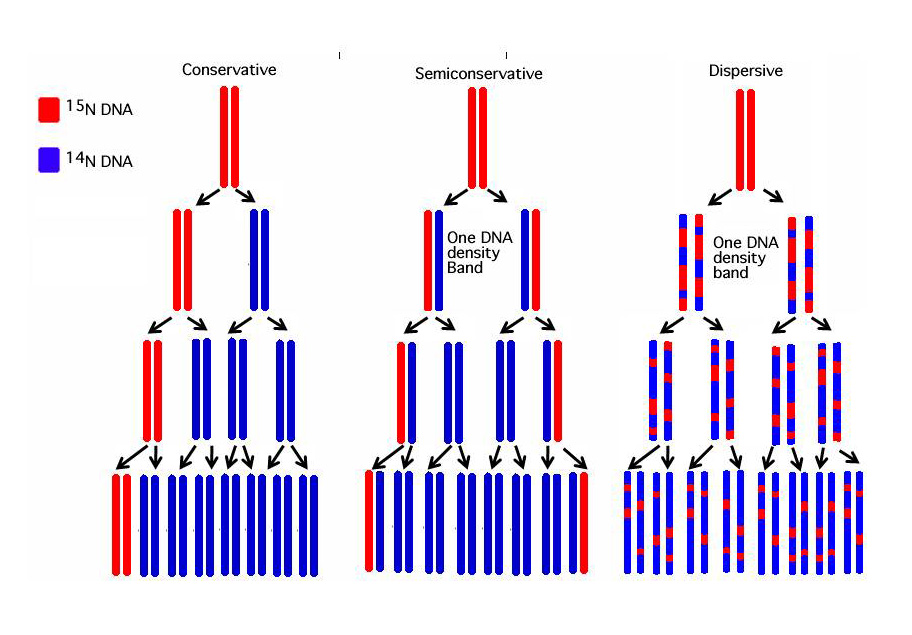
১৯৫৭ সালে ক্যালটেকের দুই গবেষক ম্যাথু মেসেলসন (Matthew Meselson) এবং ফ্রাঙ্কলিন স্টল (Franklin Stahl) নাইট্রোজেনের দুটি সমাণু বা আইসোটোপ ব্যবহার করে সেমি-কন্সার্ভেটিভ মডেলটির সত্যতা খুঁজে পান।
DNA রেপ্লিকেশনের জন্য দায়ী এনজাইমের নাম DNA-পলিমারেস। DNA-পলিমারেস প্রোক্যারিয়টিক এবং ইউক্যারিয়টিক কোষ ভেদে ভিন্ন ভিন্ন ধরণের হয়, এমনকি ইউক্যারিয়টিক কোষে একাধিক DNA-পলিমারেস এনজাইম রয়েছে যারা কার্যত একে অন্যের চেয়ে কিছুটা আলাদা। যা হোক, DNA-পলিমারেস তার কাজ শুরুর আগেই আরো কিছু প্রোটিন কাজে লেগে পরে। প্রথমে আসে হেলিকেস (Helicase) এনজাইম। হেলিকেসের কাজ হচ্ছে নিউক্লিয়টাইডের মধ্যকার হাইড্রোজেন বন্ধন খুলে ফেলা। কিন্তু, নিউক্লিয়টাইডদের খুলে রেখে দিলে তারা কিছুক্ষণ পর আবার স্বতঃস্ফূর্তভাবে জোড়া লেগে যায়। তাই হেলিকেসের পরপরই আরেক ধরণের প্রোটিন আসে যারা এই খুলে যাওয়া নিউক্লিয়টাইডদের খোলা অবস্থায় ধরে রাখে। এই প্রোটিনগুলোকে বলে সিঙ্গেল-স্ট্র্যান্ড-বাইন্ডিং-প্রোটিন (Single strand binding protein)। হেলিকেস ও সিঙ্গেল-স্ট্র্যান্ড-বাইন্ডিং-প্রোটিনের কারণে খুলে যাওয়া DNA-কে দেখায় Y-আকৃতির মতো, যাকে বলে রেপ্লিকেশন-ফোর্ক (Replication fork)।
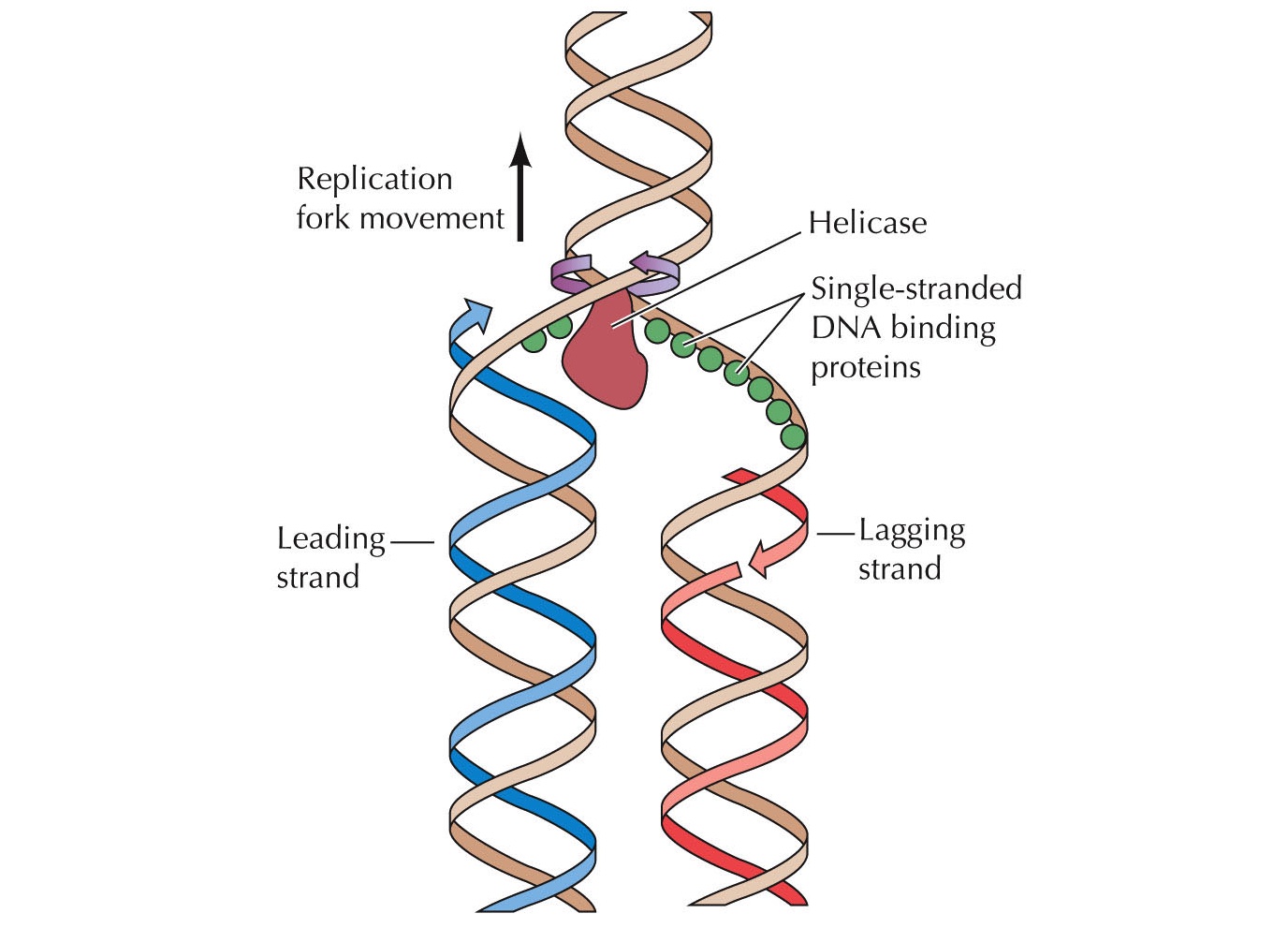
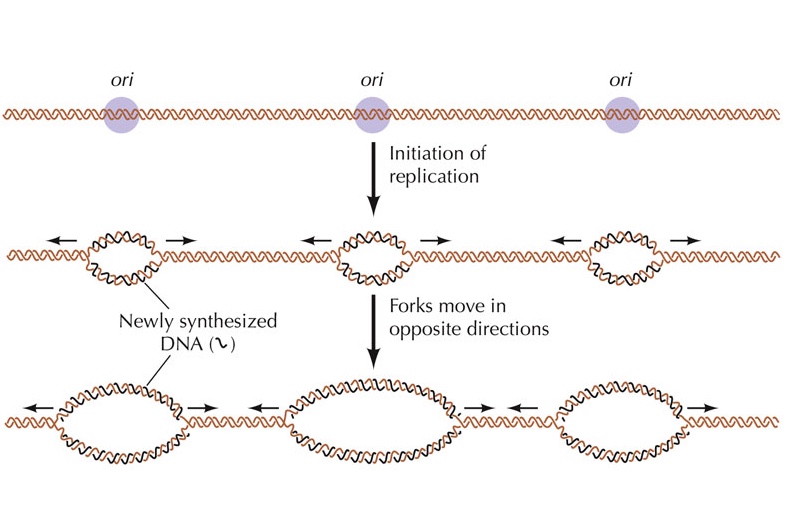
রেপ্লিকেশন শুরু হয় DNA-এর একটি নির্দিষ্ট অঞ্চলে নাম, অরিজিন-অফ-রেপ্লিকেশন বা Ori (Origin of replication)। Ori-এর নিউক্লিয়টাইডগুলোর একটা বিশেষ বিন্যাস থাকে যেটা রেপ্লিকেশন প্রক্রিয়া শুরুতে সাহায্য করে। এই নিউক্লিয়টাইডগুলো প্রোটিন সংশ্লেষণে ব্যবহৃত হয় না। প্রোক্যারিয়টিক কোষের একটি মাত্র ক্রোমোসোম যার DNA-এর দুই প্রান্ত জোড়া লেগে থাকে, ফলে প্রোক্যারিয়টিক DNA হয় বৃত্তাকার। প্রোক্যারিয়টিক DNA-এর Ori মাত্র একটি। অন্যদিকে, ইউক্যারিয়টদের ক্রোমোসোমগুলো সুদীর্ঘ এবং রেখাকৃতির। ইউক্যারিয়টিক DNA-তে একাধিক Ori থাকে, ফলে একাধিক DNA-পলিমারেস একই সাথে DNA-এর বিভিন্ন অংশের রেপ্লিকেশন চালাতে থাকে। একাধিক Ori-এর কারণে ইউক্যারিয়টিক DNA-তে একাধিক রেপ্লিকেশন-ফোর্ক তৈরি হয় যেগুলো দেখতে বুদ্বুদের মতো দেখায়, তাই এই এদের বলে রেপ্লিকেশন-বাবল (Replication bubble)।
রেপ্লিকেশন-ফোর্ক তৈরি হয়ে গেলে আশেপাশে ভেসে বেড়ানো নিউক্লিয়টাইডগুলো স্বতঃস্ফূর্তভাবে এই খোলা স্ট্র্যান্ড দুটোর উন্মুক্ত নিউক্লিয়টাইডের সাথে হাইড্রোজেন বন্ধনে আবদ্ধ হয়। অর্থাৎ, DNA-পলিমারেসকে এক এক করে নিউক্লিয়টাইড ধরে এনে খোলা স্ট্র্যান্ডে বসাতে হয় না। DNA-পলিমারেসের কাজ শুরুর আগেই নতুন স্ট্র্যান্ডের নিউক্লিয়টাইডগুলো জায়গা মতো বসে যায়। তাই, DNA-পলিমারেস কাজ হলো এই নতুন স্ট্র্যান্ডের নিউক্লিয়টাইডগুলো মধ্যে ফসফেট বন্ধন তৈরি করার মাধ্যমে ফসফেট-সুগার ব্যাকবোন তৈরি করা। ফসফেট বন্ধন শক্তিশালী কোভেলেন্ট বন্ধন, এই বন্ধন তৈরিতে শক্তির প্রয়োজন। সাধারণ নিউক্লিইক-অ্যাসিডগুলোর মাত্র একটি ফসফেট-গ্রুপ থাকে।

নতুন স্ট্র্যান্ডের নিউক্লিয়টাইডগুলোর মাঝে ফসফেট বন্ধন তৈরিতে যে শক্তির প্রয়োজন সেটি আসে ওই ভেসে বেড়ানো নিউক্লিয়টাইডগুলো থেকে। কোষে যে নিউক্লিইক-অ্যাসিডগুলো ভেসে বেড়ায় তারা একটু বিশেষ ধরণের। এদের বলে dNTP বা ডিঅক্সিনিউক্লিয়টাইড-ট্রাইফসফেট। এই নিউক্লিইক-অ্যাসিডগুলো একটির বদলে থাকে তিনটি ফসফেট-গ্রুপ (α, β, ও ૪)। এডেনিন, গুয়ানিন, সাইটোসিন, ও থায়মিনের dNTP-গুলো যথাক্রমের, ATP বা এডেনোসিন-ট্রাইফসফেট (Adenosine triphosphate), GTP বা গুয়ানোসিন-ট্রাইফসফেট (Guanosine triphosphate), CTP সাইটিডিন-ট্রাইফসফেট(Cytidine triphosphate), ও TTP থায়মিডিন-ট্রাইফসফেট (Thymidine triphosphate)। নিচের ছবি।

DNA-পলিমারেস dNTP-এর β ও ૪ ফসফেট-গ্রুপ দুটি ভেঙ্গে ফেলে, এতে যে শক্তি নির্গত হয় তা দিয়ে সে ফসফেট বন্ধন তৈরি করে। ফলে, নুতন স্ট্র্যান্ডটি তার ফসফেট-সুগার ব্যাকবোন লাভ করে।
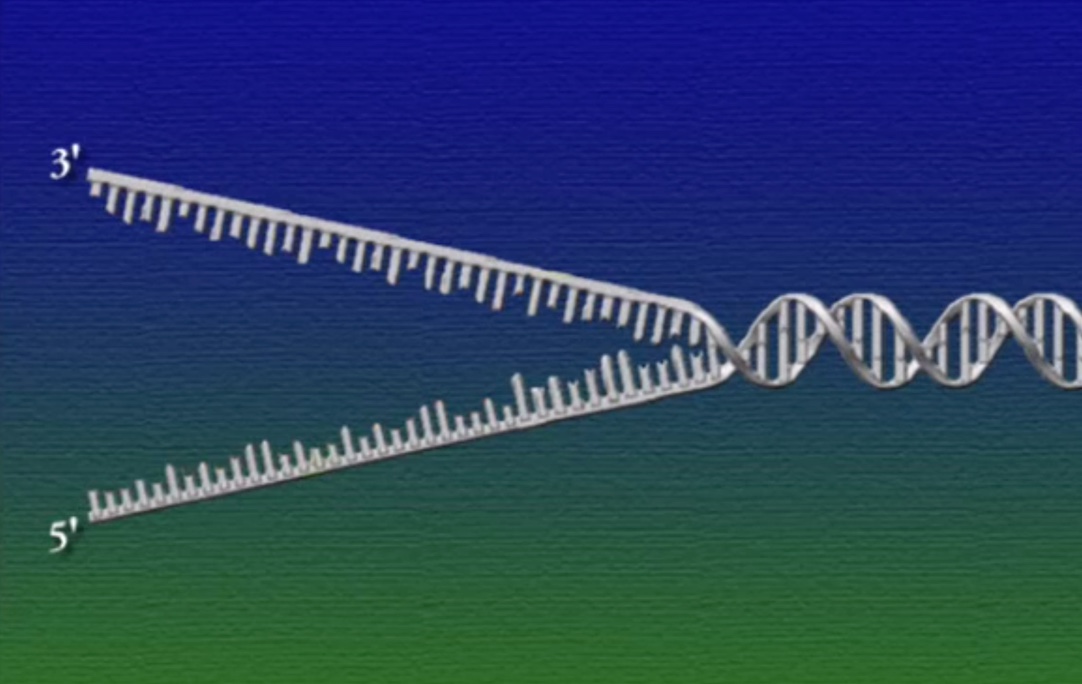
DNA-পলিমারেসের একটা সীমাবদ্ধতা হলো, এই এনজাইমের গতিপথ নির্দিষ্ট এবং একমুখী। DNA-পলিমারেস নিউক্লিয়টাইডের ৩’-প্রান্তের হাইড্রক্সিল-গ্রুপ সরিয়ে একটি ফসফেট-গ্রুপ যুক্ত করে। বলে রাখা ভালো, প্রতিটি নিউক্লিয়টাইডের ৫’-প্রান্তে এক বা একাধিক ফসফেট-গ্রুপ লাগানোই থাকে, তাই DNA-পলিমারেস এই ৫’-প্রান্ত স্পর্শ করে না (উচ্চারণ: ৩’ = থ্রি-প্রাইম, ৫’ = ফাইভ-প্রাইম)। সমস্যা হলো, DNA-তে কমপ্লিমেন্টারি স্ট্র্যান্ডগুলো বিপরীতমুখী।অর্থাৎ, একটি স্ট্র্যান্ডের মেরু ৩’—>৫’ এবং এর শুরুতেই খোলা ৩’-প্রান্ত থাকে। আর অন্যটির মেরু ৫’—>৩’ এবং এই স্ট্র্যান্ডের শুরুতে কোনো খোলা ৩’-প্রান্ত নেই। ফলে, ৫’—>৩’ স্ট্র্যান্ডটি DNA-পলিমারেসের কাজ শুরু করে রেপ্লিকেশন-ফোর্কের গোড়া থেকে।

ওপরের ছবিতে DNA-এর ডাবল-হেলিক্স কাঠামোকে সোজা করে রেললাইন মতো করে দেখানো হয়েছে। এই রেললাইনের ফসফেট-সুগার ব্যাকবোন দুটির মেরু ঠিক উল্টো। একটির ৩’ –>৫’ এবং অন্যটির ৫’—>৩’। ব্যাকবোন দুটি যুক্ত থাকে নিউক্লিয়টাইডগুলোর মধ্যকার হাইড্রোজেন বন্ধনের কারণে।

৩’—>৫’ স্ট্র্যান্ডে DNA-পলিমারেসের গতিপথের সোজা দিকে আর ৫’—>৩’ স্ট্র্যান্ডে ঠিক উল্টো দিকে।

DNA-পলিমারেস ৩’—>৫’ স্ট্র্যান্ড ধরে নির্বিঘ্নে এগিয়ে যায় এবং নতুন কমপ্লিমেন্টারি স্ট্র্যান্ড তৈরি করে (লাল রং)। এই নতুন স্ট্র্যান্ডটিকে বলে লিডিং-স্ট্র্যান্ড (Leading strand)।

DNA-পলিমারেস ৫’—>৩’ স্ট্র্যান্ডের রেপ্লিকেশন-ফোর্কের Y-কোণ থেকে শুরু করে উল্টো পথে এগুতে থাকে। ৫’—>৩’ বরাবর তৈরি হওয়া নতুন স্ট্র্যান্ডকে বলে ল্যাগিং-স্ট্র্যান্ড (Lagging strand)।

৫’—>৩’ স্ট্র্যান্ডের শেষ প্রান্তে পৌঁছানো সাথে সাথে DNA-পলিমারেস লাফ দিয়ে রেপ্লিকেশন-ফোর্কের নতুন খোলা অংশে চলে আসে এবং আবার উল্টো দিকে এগুতে থাকে। DNA-পলিমারেসের এই সোজা দিকে লাফ দেয়া এবং উল্টো দিকে এগুনোর জন্য ল্যাগিং-স্ট্র্যান্ডের মাঝে ফাঁক তৈরি হয়। জোড়া লাগার আগ পর্যন্ত এই ভাঙ্গা ল্যাগিং-স্ট্র্যান্ডগুলোকে বলে ওকাজাকি ফ্রাগমেন্ট (Okazaki fragments)।

DNA-পলিমারেস যখন পূর্ববর্তী ওকাজাকি ফ্রাগমেন্টে এসে পৌঁছে তখন সম্পূর্ণ ভিন্ন একটি প্রোটিন এসে ওকাজাকি ফ্রাগমেন্ট দুটোকে জোড়া লাগিয়ে দেয়। এবং DNA-পলিমারেস আবার রেপ্লিকেশন-ফোর্কের Y-কোণে লাফ দেয়। আর এভাবেই চলতে থাকে ৫’—>৩’ স্ট্র্যান্ডের রেপ্লিকেশন।
মানব DNA-এর নিউক্লিয়টাইড-বেসপেয়ার প্রায় ৩.২ বিলিয়ন। রেপ্লিকেশন শেষে যদি নতুন স্ট্র্যান্ডের সাথে আদি স্ট্র্যান্ডের তুলোনা করা হয় তবে রেপ্লিকেশনজনিত ভুল পাওয়া যাবে ১ বিলিয়ন বেসপেয়ারে মাত্র ১টি। এই অবিশ্বাস্য নির্ভুল রেপ্লিকেশন কিন্তু এক ধাপে হয় না। আগে বলা হয়েছে, DNA-পলিমারেস কখনোই নিউক্লিয়টাইডদের ধরে নিয়ে এসে নতুন স্ট্র্যান্ড তৈরি করে না। বরং, ভেসে বেড়ানো dNTP-রা রাসায়নিক বৈশিষ্ট্যের কারণে নিজেরাই আদি স্ট্র্যান্ডের নিউক্লিয়টাইডদের সাথে হাইড্রোজেন বন্ধনে আবদ্ধ হয়। যদি কোনো কারণে নিউক্লিয়টাইডগুলো তাদের আদি নিউক্লিয়টাইডদের কমপ্লিমেন্টারি বা পরিপূরক না হয়। অর্থাৎ, ভুলক্রমে যদি A ও T-এর বিপরীতে C বা G কিংবা, C ও G-এর বিপরীতে A বা T যুক্ত হয়। তবে, DNA-পলিমারেস যেটা করবে সেটা হলো, প্রথমে সে এই ভুল নিউক্লিয়টাইডকে ফসফেট-সুগার ব্যাকবোনে যুক্ত করবে। এরপর সে এগিয়ে যাবে পরের নিউক্লিয়টাইডে, কিন্তু, এই নিউক্লিয়টাইড জোড়া দেবার আগে সে একপ্রকার চোখ ঘুরিয়ে দেখে নেবে যে, পূর্ববর্তী বেসপেয়ারটি কমপ্লিমেন্টারি কিনা। যদি না হয় তবে, সে এক ধাপ পিছিয়ে গিয়ে ভুল নিউক্লিয়টাইডকে কেঁটে ফেলে দেবে এবং অপেক্ষা করবে কখন আরেকটি ভাসমান (সম্ভবত সঠিক) নিউক্লিয়টাইড উপস্থিত হবার জন্য। যখনই নতুন নিউক্লিয়টাইড এসে কাটা অংশে হাজির হবে, DNA-পলিমারেস সাথে সাথে বাছবিচার ছাড়াই নিউক্লিয়টাইডকে জোড়া লাগিয়ে সামনের দিকে এগিয়ে যাবে।
কোনো ধরণের সংশোধন ছাড়াই রেপ্লিকেশন জনিত ভুলের পরিমাণ অনেক বেশি, ১০হাজার বেসপেয়ারে প্রায় ১টি ভুল। DNA-পলিমারেসের কারণে এই ভুলের পরিমাণ কমে দাঁড়ায় ১০ মিলিয়ন বেসপেয়ারে ১টিতে। যা কিনা পুরো DNA রেপ্লিকেশনে প্রায় ৩০০টির ভুলের জন্য দায়ী। জাইগোট থেকে শুরু করে মানবদেহ কোষে অগণিত DNA রেপ্লিকেশন ঘটে। প্রতিবার ৩০০টি ভুল ঘটতে থাকলে সময়ের সাথে সাথে ভুলগুলো বিশাল অংকে পরিণত হবে যেটা হবে মানবদেহে জন্য অত্যন্ত ঝুঁকিপূর্ণ। তাই, কোষে জন্য DNA-পলিমারেসের এই ভুল সংশোধন প্রক্রিয়াটি যথেষ্ট নয়।
DNA রেপ্লিকেশনের সময় যেই ভুলগুলো DNA-পলিমারেস এড়াতে পারে না, সেগুলোকে সংশোধন করার জন্য থাকে একদল প্রোটিন। এদের বলা হয় মিসম্যাচ-কারেকশন-এনজাইম (Mismatch correction enzyme)। মিসম্যাচ-কারেকশন-এনজাইমগুলো DNA-পলিমারেসের পিছু পিছু এগুতে থাকে আর কমপ্লিমেন্টারি নয় এমন বেসপেয়ার খুঁজতে থাকে। পাওয়া গেলে ওই বেসপেয়ারসহ DNA-এর কিছু অংশ কেঁটে বাদ দিয়ে দেয়। এরপর ভাসমান dNTP এসে শূন্যস্থান পূরণ করে, এবং DNA-পলিমারেস এসে এই নতুন বেসপেয়ারগুলোকে ফসফেট-সুগার ব্যাকবোনে যুক্ত করে।



প্রশ্ন হলো, কোনটি ভুল নিউক্লিয়টাইড আর কোনটি সঠিক নিউক্লিয়টাইড সেটা মিসম্যাচ-কারেকশন-এনজাইমগুলো কিভাবে বুঝতে পারে? সময়ের সাথে সাথে DNA-এর রাসায়নিক পরিবর্তন ঘটে। বিশেষ করে নিউক্লিইক-অ্যাসিডগুলোর নাইট্রোজেনাস বেসগুলোতে সময়ে সাথে সাথে একাধিক মিথাইল-গ্রুপযুক্ত হতে থাকে। মিথাইল-গ্রুপ (CH3) হলো একটি কার্বন ও তিনটি হাইড্রোজেনযুক্ত একটি যৌগ। এই মিথাইল-গ্রুপ যুক্তকরণ প্রক্রিয়াকে বলে মিথাইলেশন (Methylation)। DNA-এর মিথাইলেশনের পরিমাণ সময়ের ওপর নির্ভর করে। DNA যতো পুরোনো এর মিথাইলেশনও ততো বেশি। নতুন স্ট্র্যান্ডের নিউক্লিয়টাইডগুলোর মিথাইলেশন শুরু হতে একটু সময় লাগে, প্রায় ১০ থেকে ১৫ মিনিটের মতো। মিসম্যাচ-কারেকশন-এনজাইমগুলো নিউক্লিয়টাইডগুলোর মিথাইলেশনের পরিমাণের ওপর নির্ভর করে নির্ধারণ করে কোনটি নতুন আর কোনটি পুরোনো নিউক্লিয়টাইড। বেসপেয়ার কমপ্লিমেন্টারি না হলে, মিসম্যাচ-কারেকশন-এনজাইম মিথাইলেশনবিহীন নিউক্লিয়টাইডগুলোর কেঁটে বাদ দেয়।
অবলম্বনে: “The Great Courses” থেকে প্রকাশিত “Biology: The Science of Life” by “Stephen Nowicki”